
[1] 31

\(\color{#2ECC40}s_{\color{#2ECC40}i}\) sitios de colecta con coordenadas geográficas (longitud. latitud).
\(\color{#2ECC40}D\) area de estudio (zona metropolitana de Guadalajara).
\(\color{#2ECC40}Y_{\color{#2ECC40}{si}}\) es la variable de respuesta (Número de Huevos por Ovitrampa o Manzana).
\(\color{#2ECC40}Y_{\color{#2ECC40}{si}}\) tiene una distribución (binomial negativa ó zibn).
\(\color{#2ECC40}U_{\color{#2ECC40}{si}}\) el efecto espacial & el proceso ocurre en un campo gaussiano continuo (Gaussian Field).
Modelo sin correlación espacial \[\begin{align*} Huevos & = \beta{_0} + \beta x + \epsilon{_i} \\ \beta{_0} & = intercepto \\ \beta x & = pendiente \\ \epsilon{_i} & = error \\ \epsilon{_i} &\sim {\sf Norm}(0, \sigma^2) \\ \end{align*}\]
Modelo con efecto espacial \[\begin{align*} Huevos & = \beta{_0} + \beta x + u{_i}+\epsilon{_i} \\ Huevos & = \beta{_0} + u{_i}+\epsilon{_i} \\ u{_i} & = efecto espacial \\ u{_i} &\sim {\sf GF}(0, \sum) \\ \end{align*}\]
Cuando D es regular
Se usa Gausian Makovian RF (GMRF) para aproximar la matriz de covarianzas
Cuando D es irregular
Se usa SPDE & Elemento Finito para aproximar la matriz de covarianzas
SPDE
\[(K^2-\Delta)^{a/2}U(s) = W(s)\]
Elemento Finito
\[u(s) = \sum_{i=1}^{G} \alpha{_k}(s{_i})W{_k}\]

[1] 31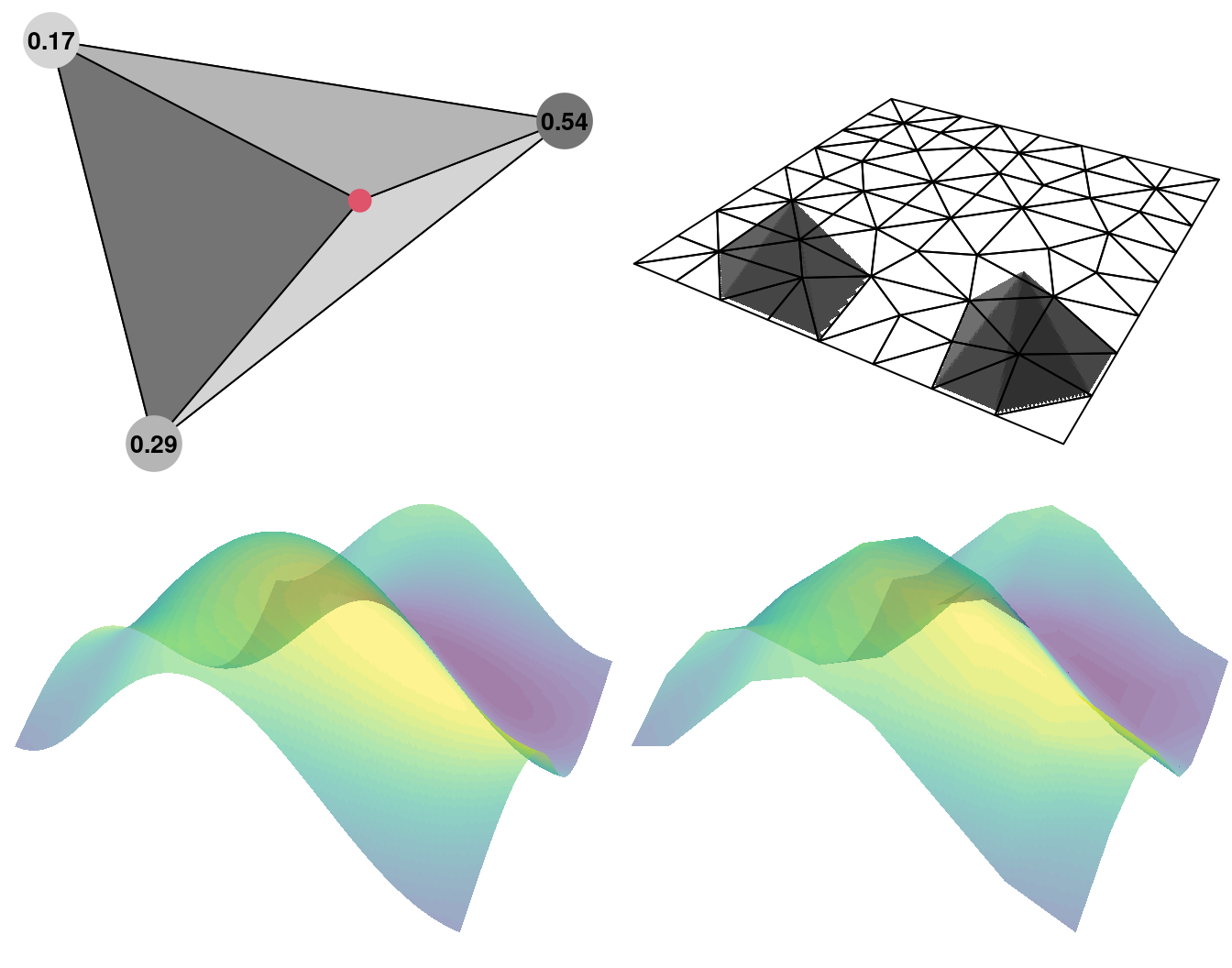
Coordenadas en los vértices
Sí \(s{_i}\) caé en el vértice 1, el resto 0. \(u(s) = w{_k}\)
Cordenadas dentro del Triángulo
Sí \(s{_i}\) caé dentro del triángulo se ponderan los tres puntos \[u(s) = \sum_{i=1}^{G} \alpha{_k}(s{_i})W{_k}\]
\[0.17 * W{_1} + 0.54 * W{_3} + 0.29 * W{_3}\]
Coordenadas sobre un borde
Sí \(s{_i}\) caé en el borde se ponderan los dos valores
\[u(s) = \sum_{i=1}^{G} \alpha{_k}(s{_i})W{_k}\]

En analisis geoestadístico de la vigilancia entomológica con ovitrampas se realiza con el paquete deneggs. El paquete es parte del dengueverse & tiene tres funciones para realizar la predicción del número de huevos en áreas donde no se colectó de una localidad.
La función spde_pred_map tiene la característica de realizar el analisis geoestadístico por semana con siete diferentes distribuciones (poisson, zeroinflatedpoisson0, zeroinflatedpoisson1, nbinomial, nbinomial2, zeroinflatednbinomial0, & zeroinflatednbinomial1). La función eggs_hotspots realiza el análisis por semana con solo una distribución y la función eggs_hotspots_week realiza el análisis para todas las semanas del año.
path_ovitraps <- "/Users/fdzul/Library/CloudStorage/OneDrive-Personal/datasets/CENAPRECE/2023/14_jalisco"
path_coord <- paste(path_ovitraps,"DescargaOvitrampasMesFco.txt", sep = "/" )
x <- deneggs::eggs_hotspots(path_lect = path_ovitraps,
cve_ent = "14",
locality = c("Guadalajara", "Zapopan",
"Tlaquepaque", "Tonalá"),
path_coord = path_coord,
longitude = "Pocision_X",
latitude = "Pocision_Y",
aproximation = "gaussian",
integration = "eb",
fam = "zeroinflatednbinomial1",
k = 80,
palette_vir = "magma",
leg_title = "Huevos",
plot = FALSE,
hist_dataset = FALSE,
sem = 41,
var = "eggs",
cell_size = 3000,
alpha = .99)
# Step 1. define the path ####
path_ovitraps <- "/Users/fdzul/Dropbox/CENAPRECE/2024/31_yucatan"
path_coordinates <- paste(path_ovitraps, "DescargaOvitrampasMesFco.txt", sep = "/")
# Step 2. run the function ####
library(magrittr)
library(sf)
x <- deneggs::spde_pred_map(path_lect = path_ovitraps,
path_coord = path_coordinates,
cve_ent = "31",
locality = "Merida",
longitude = "Pocision_X",
latitude = "Pocision_Y",
aproximation = "gaussian",
integration = "eb",
k = 30,
plot = TRUE,
week = 4,
var = "Huevecillos",
cell_size = 2000,
alpha = .99,
palette_vir = "magma",
leg_title = "Huevos")
email : felipe.dzul.m@gmail.com
celular : 9999580167
slides: https://calm-hummingbird-41cb33.netlify.app/talks/hotspots_eggs_R/#/